用聚苯乙烯-共-丙烯酸复合纳米球检测霍乱弧菌 DNA 的超灵敏生物传感器
摘要
一种用于检测致病性霍乱弧菌的超灵敏电化学生物传感器 (V . 霍乱 ) DNA 是基于聚苯乙烯-共聚丙烯酸 (PSA) 乳胶纳米球-金纳米粒子复合材料 (PSA-AuNPs) DNA 载体基质开发的。使用电活性蒽醌寡核苷酸标记的微分脉冲伏安法 (DPV) 用于测量生物传感器响应。在 DNA 乳胶粒子电极上加载金纳米粒子 (AuNPs) 显着放大了 DNA 杂交的法拉第电流。连同已报道的探针的使用,生物传感器已表现出高灵敏度。 DNA 生物传感器对目标 DNA 产生了可重现的宽线性响应范围,从 1.0 × 10 −21 到 1.0 × 10 −8 M(相对标准偏差,RSD =4.5%,n =5) 检测限 (LOD) 为 1.0 × 10 −21 M (R 2 =0.99)。生物传感器在 91% 到 109% (n =3) 用于检测 V . 霍乱 加标样品中的 DNA,可重复用于六次连续 DNA 检测,重复性 RSD 值为 5% (n =5)。电化学生物传感器的响应在长达 58 天的储存期内稳定并保持在其原始响应的 95%。
背景
霍乱弧菌 是一种食源性病原体,可通过急性水样腹泻在人类中引起霍乱流行。霍乱疫情在世界某些地区仍然是一个严重的问题,例如亚洲和非洲,并导致社会经济地位低下 [1,2,3,4]。这种肠道病原体是发病率和死亡率的主要原因,尤其是在发展中国家 [5]。各地区霍乱的流行和大流行主要是由V.霍乱 血清群 O1 和 O139 [1, 2, 6]。 V。霍乱 血清群 O1 有两种主要血清型,即稻叶和小川,它们可能在霍乱流行之间交替出现。第三种血清型 Hikojima 也存在,但罕见且不稳定。负责 O1 抗原生物合成的基因被指定为 rfb。定义血清型 Inaba 和 Ogawa 的突变是 rfbT 基因中的单个缺失突变 [7]。然而,据报道,严重腹泻的人类偶发的食源性疾病暴发也是由非 O1/非 O139 V 引起的。霍乱 通过摄入未煮熟的海鲜 [8] 或暴露于受污染的水生环境 [9]。 V的第一次流行。霍乱 O139 于 1992 年在孟加拉国和印度发生,然后迅速传播到东南亚其他国家[10]。 2005年,全世界向世界卫生组织(WHO)报告的霍乱病例总数为131,943例,死亡2272例[11]。
寻求监测或诊断产毒 V 的有效方法。霍乱 细菌对于控制霍乱流行势在必行。 V 的传统鉴定。霍乱 通常通过分离和筛选细菌来实现,其中包括在碱性蛋白胨水 (APW) 中进行预富集,然后分离 V。霍乱 在硫代硫酸盐柠檬酸盐胆盐蔗糖琼脂(TCBS)培养基上的检测和特异性抗血清的玻片凝集试验鉴定[12]。然而,这种技术非常耗时和劳动密集型,几天后获得的结果将意味着延迟临床诊断和患者治疗。用于检测 V 的涉及 PCR 扩增的分子方法。霍乱 [13] 减少了诊断时间 [14],但 PCR 方法需要熟练的专业和昂贵的基础设施,这在资源设置较低的国家难以实施。据报道,基于免疫层析原理的快速诊断测试可用于不连续或同时检测 V。霍乱 血清群 O1 和 O139。用于检测 V 的其他一些基于免疫测定的技术。霍乱 如酶联免疫吸附试验(ELISA)、凝集、免疫荧光和石英晶体微量天平(QCM)。然而,这些技术中的大多数都需要复杂的仪器、较长的分析时间和具有详细技术知识的高素质人员 [15,16,17,18,19,20]。
电化学方法由于其灵敏度高、特异性强、操作简单、方案经济、检测速度快、与微加工技术兼容等优点,在核酸检测中引起了广泛关注[21, 22]。此外,结合小型化技术的电化学方法可用于原位分散分析,例如基于微流控芯片的DNA生物传感器装置,这对于实际设置非常有用[23]。电化学测量中使用的电极种类繁多,例如玻碳电极(GCE)、碳糊电极(CPE)、金电极和铂电极。最近,由于丝网印刷电极 (SPE) 具有一些独特的特性,例如提供低背景电流和宽电位窗口,碳墨水价格低廉,因此具有成本效益,并且可以大规模生产,因此研究集中在使用丝网印刷电极 (SPE) 上。 .
有几种电化学方法可用于检测 V。霍乱 由一系列复杂的步骤组成。电化学V。霍乱 Liew 等人报道的基因传感器。 [24] 使用电化学吸附方法将 DNA 探针固定在碳 SPE 上。冻干的 AuNPs 改性多层 PSA 颗粒与聚电解质形成生物共轭物与亲和素,在夹心 DNA 杂交试验中充当报告标记。然而,需要添加山梨糖醇稳定剂来保存 PSA-AuNPs-亲和素生物共轭物,以将 DNA 生物传感器的运行时间延长至 30 天。酶电化学V。霍乱 Yu 等人最近设计了 DNA 生物传感器。 [25],其中硫醇化抗荧光素偶联碱性磷酸酶(抗 FCAP)标记的 DNA 探针通过金硫醇化学与金 SPE 结合。目标 DNA 用通用荧光素标记,以允许通过酶促将 α-萘基磷酸酯转化为具有电活性的 α-萘酚来实现 DNA 杂交识别。然而,该检测方案需要大约 95 分钟的较长时间进行 DNA 杂交,用功能性酶标记 DNA 杂交体,然后在进行安培测量之前将电极在无电活性的 α-萘基磷酸酯底物中孵育。 Low 及其团队成员开发了另一种酶促电化学 DNA 生物传感器,该传感器基于与生物素化 DNA 探针结合的抗生物素蛋白涂层碳 SPE [26]。在这种双杂交策略中也使用了地高辛 (DIG) 标记的报告探针,它位于 cDNA 序列的两侧。辣根过氧化物酶连接的抗 DIG(抗 DIG-HRP)被用作电化学标记,它可以同时催化 3,3',5,5'-四甲基联苯胺(TMB)的氧化和 H2O2 的还原,产生电子转移到电极表面,用于 DNA 杂交事件的电化学转导。 Patel 等人描述了一种基于硫醇化 DNA 探针固定镀金玻璃电极的简便 DNA 生物传感器设计。 [22] 用于V 的快速检测。霍乱 , 亚甲蓝用作 DNA 杂交指示剂。然而,该系统的线性检测范围仅限于μM水平,限制了其在临床样品中的应用。
乳胶-金纳米颗粒以前曾被用作 DNA 杂交标记,通过抗生物素蛋白/生物素与 DNA 探针结合来检测 V。霍乱 [24]、鱼类病原体入侵隐孢子菌 [27],E。大肠杆菌 [28] 和非特异性 DNA 杂交 [29],其中乳胶球涂有多层聚电解质,然后金纳米粒子的带负电荷的胶体通过静电附着在其上。 Kawde 和 Wang [29] 将 PSA 乳胶颗粒连接到 DNA 报告探针上,通过加载带有生物素涂层 AuNP 的链霉亲和素涂层乳胶颗粒用作 DNA 杂交标记。关等人。 [24, 27] 和 Liew 等人。 [24, 27] 还报道了使用金-PSA-DNA 探针偶联物的相同抗生物素蛋白-生物素结合方法。皮尼苏万等人。 [28] 报道了使用静电方法加载附着在 DNA 报告探针上的 PSA 颗粒,并将聚电解质金包被的 PSA 颗粒用作杂交标记,放大 DPV 电流响应。
在这项研究中,我们报告了一种不同的 DNA 固定方法,使用乳胶金纳米粒子作为 DNA 探针固定基质,以开发一种高灵敏度的 V 检测系统。霍乱 脱氧核糖核酸。使用 1-乙基-3-(3-二甲氨基丙基)碳二亚胺盐酸盐/N-羟基琥珀酰亚胺(EDC/NHS)化学作为偶联剂,以提高羧化胶乳的固定效率 [30],以非常简单和快速的程序进行 DNA 固定颗粒表面。 DNA 杂交检测基于夹心型分析,其中涉及固定的 DNA 探针和目标序列之间的杂交反应,然后是信号/报告探针。蒽醌-2-磺酸一水合物钠盐(AQMS)被用作电化学标记来监测杂交事件。所提出的亚微米级乳胶颗粒提高了 DNA 探针的结合能力,并且通过掺入高导电金纳米颗粒 (AuNPs) 提高了 DNA 生物传感器的灵敏度。 DNA 生物传感器对 V 的检测表现出非凡的灵敏度。霍乱 与目前报道的亲和素-生物素技术相比,cDNA 和极低的 zeptomolar 水平检测限 [24, 26]。
方法
化学品和试剂
苯乙烯 (St) 和丙烯酸 (AA) 购自 Fluka。 HAuCl4·3H2O、无水柠檬酸三钠、十二烷基硫酸钠 (SDS)、1-乙基-3-(3-二甲氨基丙基)碳二亚胺盐酸盐 (EDC) 和 N-羟基琥珀酰亚胺 (NHS) 购自 Sigma-Aldrich。过硫酸铵 (APS)、氢溴酸和溴分别由 Riedel-De Haën、Ajax Finechem 和 Panreac 提供。所有化学溶液均用去离子水配制。 30 碱基目标和错配合成寡核苷酸均从 Bio Service Unit (NSTDA) 采购。非互补 DNA (ncDNA) 和信号探针来自 Sigma,5'-氨基修饰的捕获探针来自 Bioneer。捕获探针在 0.05 M 磷酸钾缓冲液 (pH 7) 中制备,而目标 DNA、错配目标、报告探针和非互补 DNA 溶液在磷酸钠缓冲液 (0.05 M,pH 7) 中制备。本研究中使用的寡核苷酸序列见表1。
设备
使用配备 GPES (4.0.007) 软件的恒电位仪/恒电流仪 (Autolab PGSTAT12, Metrohm) 进行电分析测量。伏安实验是用传统的三电极系统进行的,该系统由碳糊丝网印刷工作电极 (SPE) (Quasense, Bangkok, Thailand)、Ag/AgCl 参比电极 (3 M 的 KCl) 和铂棒 ( 2 毫米直径)对电极。差分脉冲伏安法 (DPV) 技术用于所有电化学研究,在 0.02 V 步进电位和 0.5 V/s 扫描速率下,在 0.0 至 +1.0 V 的 4.5 mL 测量缓冲液(0.05 M 磷酸钾缓冲液)中,pH 7 和环境温度。本研究中测量的所有电位均参考 Ag/AgCl 电极,并使用声波器浴 (Elma S30H) 制备均匀溶液。扫描电子显微镜(SEM,LEO 1450VP)用于测定乳胶球的尺寸和分布。
方法
胶体金纳米粒子的合成
按照Turkevich方法[31]通过柠檬酸钠还原制备胶体AuNP。简而言之,将约 10 mL 的 5 mM HAuCl4·3H2O 溶解在 180 mL 去离子水中,并在组合的热板磁力搅拌器装置上在连续搅拌条件下煮沸。十毫升 0.5% (w /v )的柠檬酸三钠加入沸腾的溶液中,观察溶液的颜色由浅红色逐渐变为红宝石红色。
乳胶粒子的制备
如Polpanich等人所述,通过无皂乳液共聚反应制备胶乳颗粒。 [23] 进行了一些修改。简而言之,将约 190 g 的去离子水在三颈烧瓶中用氮气吹扫,该烧瓶浸没在水浴中约 1 小时,在 350 rpm 的搅拌下进行。然后加入 20 克 St 和 0.5 克 AA,并将温度保持在 70°C。之后,将约0.2 g APS加入10 mL去离子水中,然后倒入三颈烧瓶中的制剂中以引发自由基聚合反应,聚合过程进行8 h。通过用去离子水以 13,000 rpm 离心两次 20 分钟 [23, 27, 28] 来收集合成的羧基胶乳球,并在室温(25°C)下重新悬浮在去离子水中直至使用。 PSA胶乳颗粒的形貌和平均粒径通过扫描电子显微镜(SEM)测定。
修饰 SPE 表面和 DNA 探针固定
在表面改性之前,用去离子水彻底冲洗碳 SPE,然后滴涂 3 mg/mL 的 PSA 悬浮液,并在环境条件下风干,然后滴涂 5 mg/mL 胶体 AuNP .用CV方法检查了用乳胶颗粒和AuNP改性前后碳SPE的电化学特性。然后将乳胶-AuNPs 改性的碳 SPE(PSA-AuNPs-SPE)用去离子水冲洗并浸入 0.1 M 磷酸钾缓冲液(pH 5)中,该缓冲液含有碳二亚胺交联剂,即 0.002 M EDC 和 0.005 M NHS 2 小时 [32],然后在含有 5 μM 捕获探针的 0.05 M 磷酸钾缓冲液 (pH 7) 中浸泡过夜。之后,将 DNA 修饰的 PSA-AuNPs-SPE(DNA-PSA-AuNPs-SPE)用磷酸钾缓冲液(0.05 M,pH 7)彻底清洗,以去除物理吸附的探针。将 DNA 电极浸入 0.05 M pH 7 的磷酸钠缓冲液中,其中含有线性靶 DNA (1 μM) 和 AQMS DNA 杂交标记 (5 mM) 进行部分杂交 1 小时,然后浸入 0.05 M 磷酸钠缓冲液(pH 7) 用 1 μM 报告探针和 5 mM AQMS 再调节 1 小时,以进行完整的 DNA 杂交过程。最后,用磷酸钾缓冲液 (0.05 M,pH 7) 冲洗 DNA 电极以去除未杂交的寡核苷酸序列。用 DPV 方法研究了附着在 SPE 上的每种扩展物质的电化学响应。图 1 表示开发 V 的逐步程序。霍乱 基于胶体PSA-AuNPs固相载体的DNA生物传感器。
<图片>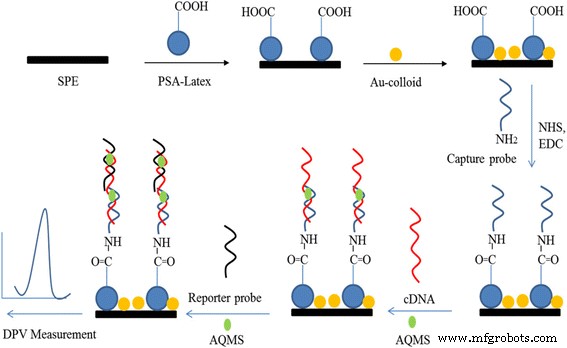
DNA生物传感器分步制作流程示意图
合成寡核苷酸杂交的优化
在确定 DNA 的动态线性范围之前,已经检查了各种参数(例如 DNA 探针和 AQMS 浓度、pH、离子强度和缓冲容量)对固定 DNA 探针与信号探针和目标 DNA 的杂交响应的影响生物传感器。此外,在开发 V 之前还评估了捕获探针固定和 DNA 杂交持续时间以及生物传感器寿命和再生。霍乱 DNA 生物传感器已准备好用于加标和回收实验。捕获探针和 AQMS 的负载通过将它们的浓度分别从 1 到 6 μM 和 0.1-5 mM 变化进行优化,而目标 DNA 和报告探针的浓度在 0.05 M 磷酸钠缓冲液 (pH 7.0) 中保持在 5 μM。通过将磷酸钠缓冲液的 pH 值和浓度分别从 pH 5.5 改为 8.0 和将 0.001 改为 1.000 M,进行 pH 影响和缓冲液浓度研究。电化学DNA生物传感器DNA杂交响应中不同阳离子的存在通过添加Na + , K + , Ca 2+ , 和 Fe 3+ 将 1.0 M 离子注入含有 1 mM AQMS 和 5 μM cDNA 和 pH 7.0 检测探针的 DNA 杂交缓冲液中。通过在 pH 7.0 下在 0.1-3.0 M 范围内改变 NaCl 浓度来检查离子强度效应。 DNA生物传感器的动态范围在1.0 × 10 -21 到 1.0 × 10 −8 MV。霍乱 使用 5 μM 和 pH 7.0 的恒定信号探针浓度的 cDNA。 DNA 探针固定持续时间通过将 DNA 电极浸入 5 μM 捕获探针溶液(pH 7.0)中 1 到 13 小时来确定,并且每 1 到 2 小时采集一次 DPV 响应。同时,通过允许 DNA 杂交反应发生在 15 到 180 分钟之间来确定 DNA 杂交时间,并且每 15 到 30 分钟记录一次 DNA 生物传感器响应。 DNA 生物传感器的保质期是通过定期测量 DNA 生物传感器对 5 μM V 检测的响应来确定的。霍乱 cDNA 120 天。对于每个夹心杂交测定,使用新的 DNA 电极重复进行 5 次分析。 DNA 电极的再生使用 0.1 M NaOH 溶液进行 4 分钟,DNA 生物传感器的再杂交(60 分钟)使用含有 5 μM cDNA 和检测探针以及 1 mM AQMS 的再杂交溶液在 2.0 M 离子强度下完成在 0.05 M 磷酸钾缓冲液 (pH 7.0) 中。再生实验一式六次。
V.霍乱 使用基于 PSA-AuNPs 的电化学 DNA 生物传感器进行定量
各种V。霍乱 细菌菌株,即 J2126-I、J2126-II、J3324-I、J3324-II、J3330-I、J3330-II、CDHI 5294-II 和 UVC1324,包括弗氏柠檬酸杆菌 (CF-I) 和 弗氏柠檬酸杆菌 (CF-II) 来自吉打 AIMST 大学应用科学学院微生物实验室。然后使用 QIAGEN Genomic-tip 500/G 根据制造商的方案对这些细菌进行基因组 DNA 提取。然后使用磷酸钠缓冲液 (0.05 M, pH 7.0) 将提取的 DNA 稀释 100 倍。将约 300 mL 提取的含有 2.0 M NaCl 和 1 mM AQMS 的 DNA 超声 15 分钟以释放 DNA 断裂。然后,将固定的 DNA 探针浸泡 1 小时以进行 DNA 杂交过程,并用 0.05 M 磷酸钾缓冲液 (pH 7.0) 小心洗涤以去除未结合的 DNA。测量基于 DPV 峰值电流的 DNA 生物传感器响应的评估,并将其与 DNA 电极产生的电流响应进行比较,而未与 cDNA 反应作为控制信号。每个实验在相同的实验条件下一式三份进行。常见的t 测试用于确定 DNA 生物传感器响应与对照响应之间的显着差异。 V 的恢复。霍乱 J3324 和 V。霍乱 UVC1324 DNAs at 1.0 × 10 −4 μg μL −1 , 1.0 × 10 −5 μg μL −1 , 和 1.0 × 10 −6 μg μL −1 然后使用所提出的基于 PSA-AuNPs 的电化学 DNA 生物传感器将其掺入杂交缓冲液中。
结果与讨论
乳胶颗粒的形态和乳胶-金纳米颗粒改性 SPE 的电化学行为。
图 2 显示了平均粒径为 186.1 ± 4.6 nm 的羧化胶乳球的 SEM 显微照片。 PSA 球体的均匀尺寸分布允许将 DNA 分子均匀固定在乳胶表面,以提高 DNA 生物传感器的再现性响应。扫描电子显微镜(SEM,LEO 1450VP)用于测定乳胶球的尺寸和分布。
<图片>
合成后的 PSA 胶乳球体放大 10,000 倍的 SEM 显微照片
修改后的 SPE 的电动力学结果列于表 2 中。峰电位分离 (ΔEp) 表示系统的电子转移动力学。由于胶体共聚物颗粒层中的缓慢电荷转移过程,PSA改性的SPE(PSA-SPE)显示出最高的ΔEp值,这使得系统向准可逆状态移动。然而,当AuNPs负载在PSA-SPE上时,ΔEp的减少意味着电极表面电子转移速率的提高。
图 3 显示了胶乳改性 SPE 上 AQMS 氧化响应的差分脉冲伏安图和 V 的顺序杂交响应。霍乱 DNA生物传感器。在仅含有乳胶修饰微球和不含固定捕获 DNA 的电极(实验 (a) 和 (b))和在存在 cDNA 和报告探针的情况下用固定捕获 DNA 探针修饰的电极之间观察到显着的 DPV 峰值电流差异(实验(g ))。这表明胺化 DNA 捕获探针通过 EDC/NHS 偶联方案成功固定在涂覆的羧化 PSA 乳胶球上 [33]。实验 (g) 还显示,与 DNA 电极相比,在存在 ncDNA 和报告的报告探针(实验 (d))、错配的 DNA 和报告的报告探针(实验 (e))以及目标 cDNA 的情况下,DPV 响应要高得多没有报告探针(实验(f))。这是由于靶 DNA 与捕获和报告探针通过 DNA 生物传感器表面上的夹心杂交反应完全杂交,如实验 (g) 所示。这也表明使用报道的探针可以增强来自 DNA 杂交的信号。尽管如此,在靶 DNA 存在下观察到的杂交产生的 DPV 电流(实验(f))仍然高于未杂交 DNA 观察到的 DPV 电流信号(实验(c)、(d)和(e)).
<图片>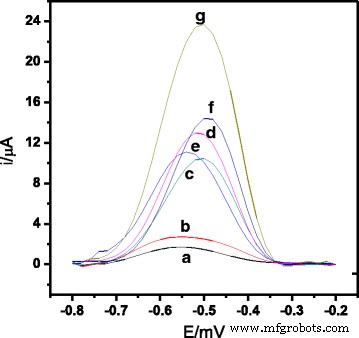
来自电极 AQMS 的差分脉冲伏安图信号 (a ) PSA-SPE, (b ) PSA-AuNPs-SPE, (c ) 捕获探针-PSA-AuNPs-SPE 并且当存在 (d ) ncDNA 和报告探针,(e ) 错配 DNA 和报告探针, (f ) 单独的 cDNA,和 (g ) cDNA 和报告探针
DNA 探针加载和 AQMS 浓度的影响
通过 AQMS 电化学氧化反应观察 DNA 探针浓度对 DNA 杂交反应的影响。图 4a 显示 DNA 生物传感器响应随着固定在 PSA-AuNPs-SPE 上的 DNA 探针量从 1 到 4 μM 的增加而逐渐增加。这归因于插入双链 DNA (dsDNA) 的电活性 AQMS 的数量增加,以通过固定的 DNA 螺旋实现电子转移。观察到 DNA 生物传感器的 DPV 响应在 4 到 6 μM DNA 探针之间几乎趋于平稳,这表明在电极表面实现了最佳 DNA 探针负载 [34]。因此,在随后的实验中,选择 4 μM 捕获探针作为最佳 DNA 探针加载。 AQMS 标记的浓度也在 0.1 至 5.0 mM 的测量电解质中进行了优化,发现 1 mM 的 AQMS 浓度足以实现最佳的 DNA 嵌入反应(图 4b)。
<图片>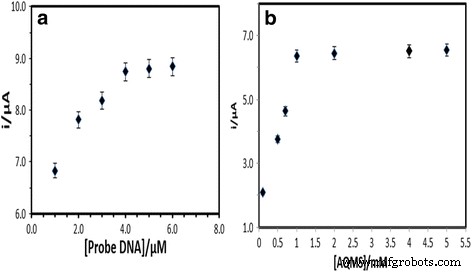
捕获探针(a ) 和 AQMS 浓度 (b ) 在 0.05 M 磷酸钠缓冲液 (pH 7.0) 中使用 5 μM cDNA 和信号探针进行的 DNA 生物传感器响应
pH、离子强度和缓冲容量的影响
DNA 杂交反应的速率很大程度上取决于溶液的 pH 值。从图 5a 中可以看出,在更酸性的环境下,DNA 磷酸二酯骨架的质子化降低了 DNA 分子的溶解度,最终降低了 DNA 杂交反应速率。而在碱性介质中,它打破了将 DNA 碱基对结合在一起的弱氢键。最佳 DNA 杂交反应在中性条件下更有利,从而促进更多捕获探针与目标 DNA 杂交,随后允许插入 AQMS 氧化还原探针,使 DNA 杂交识别成为业务。因此,pH 7.0 的 0.05 M 磷酸钠缓冲液用作 DNA 杂交介质,用于随后的 DNA 生物传感器研究。带正电的离子,例如 Ca 2+ , Na + , K + , 和 Fe 3+ 离子可以与带负电荷的 DNA 磷酸二酯链相互作用。这种离子反应会中和 DNA 分子的电荷,从而降低 DNA 分子之间的空间排斥,从而缓和 DNA 杂交反应。图 5b 描绘了一些阳离子对 DNA 杂交反应的影响。注意到 DNA 杂交反应在 Na + 顺序的正电荷离子存在下增加> K + > Fe 3+ > Ca 2+ . Ca 2+ 和 Fe 3+ 由于 Ca 2+ 的离子相互作用,发现离子可显着降低 DNA 杂交反应 和 Fe 3+ 离子与来自缓冲溶液的磷酸盐离子,从而导致形成不溶性磷酸盐化合物。这降低了介质的离子含量,从而增加了 DNA 分子之间的静电排斥。在 Na + 存在下获得最高的 DNA 杂交电流 离子,因为与 K + 相比,它的尺寸更小,对 DNA 糖-磷酸骨架的亲和力更强 离子克服DNA带负电荷的磷酸基团之间的空间位阻和静电排斥。
<图片>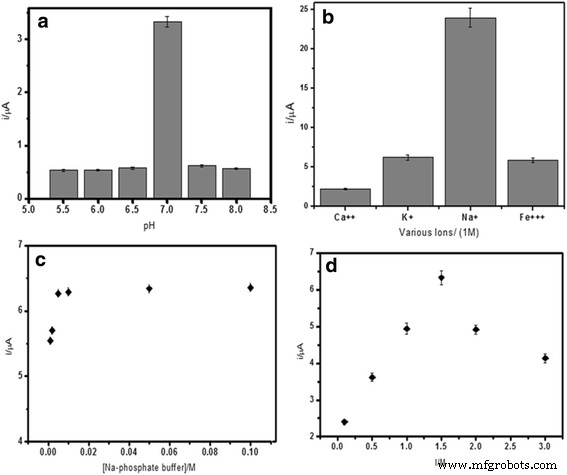
pH 的影响 (a ) 各种阳离子 (b ), 缓冲液浓度 (c ) 和离子强度 (d ) 关于电化学 V 的 DNA 杂交响应。霍乱 DNA生物传感器。用 5 μM cDNA 和报告探针进行杂交,然后插入 1 mM AQMS
此外,溶液的离子强度也会影响 DNA 生物传感器的响应。图 5c、d 显示了最佳缓冲容量,离子强度是使用 0.05 M 磷酸钠缓冲液实现的,pH 值分别固定在 pH 7.0 和 2.0 M NaCl。在这种情况下,它最有利于DNA杂交反应;因此,产生了高 DPV 响应。在溶液的最佳缓冲容量和离子强度下,DNA 分子之间的静电排斥降低,从而改善 DNA 杂交反应。相反,当离子含量过低或过高时,位阻和静电排斥占主导地位,限制了DNA分子的杂交。
建立V。霍乱 DNA生物传感器校准曲线
从图 6a 所示的结果可以看出,DNA 生物传感器响应随着 cDNA 浓度的增加成比例增加,从 1.0 × 10 -21 到 1.0 × 10 −8 M (R 2 =0.99) 检测限为 1.0 × 10 −21 M. 检测限的计算基于生物传感器响应在响应曲线上的标准偏差的三倍,近似检测限除以线性校准斜率。 DNA 生物传感器的广泛线性检测范围是由于亚微米范围内高度单分散和球形的 PSA 乳胶颗粒用作 DNA 固定的载体基质。乳胶颗粒表面上的富含丙烯酸的层为 DNA 捕获探针的附着提供了一个大的结合位点,以创建一个被 DNA 接受层覆盖的最大表面。此外,在PSA修饰的SPE上掺入AuNP进一步放大了DNA杂交响应的分析信号,这使得DNA生物传感器具有高灵敏度(图6b)。
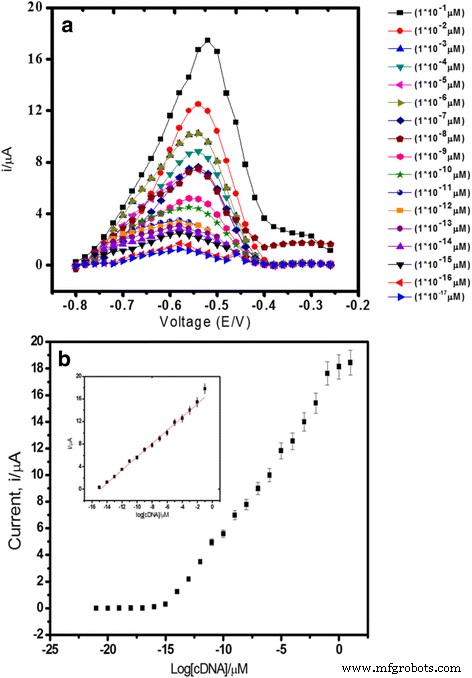
Differential pulse voltammograms (a ) and DNA biosensor linear range (b ) obtained using various cDNA concentrations from 1.0 × 10 −15 to 1.0 × 10 −1 μM V. cholerae target DNA and 5 μM signal probe
DNA Probe Immobilisation and Hybridization Times
It took about 8 h for the capture probe to be immobilised on the PSA copolymer particles surface, as illustrated by the DNA biosensor response in Fig. 7a, which showed a current increment from 1.0 to 8.0 h of capture probe immobilisation time, after which no obvious change in the DPV current was observed. Longer immobilisation time resulted in a higher amount of DNA probes immobilised onto the latex. After 8.0 h of exposure to the DNA probes, the hydrophilic functional latex with reactive carboxyl groups at the surface was presumably fully attached with the DNA probes. DNA hybridisation time, on the other hand, is the rate limiting step, which determines the response time of the DNA biosensor. Based on the DNA biosensor response trend in Fig. 7b, the response time of the V. cholerae DNA biosensor developed in this study was estimated to be about 60 min for the dual hybridisation processes to complete.
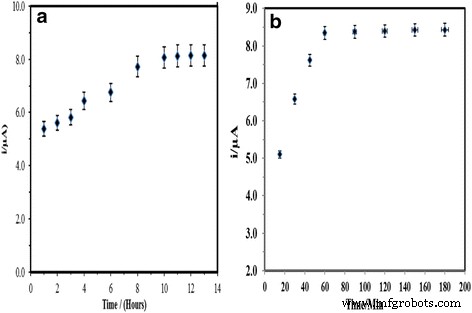
DNA probe immobilisation duration on the immobilised PSA latex colloidal particles (a ) and DNA hybridization duration of the DNA biosensor (b ) in 0.05 M potassium phosphate buffer at pH 7.0 containing 5 μM target DNA and reporter probe and 1 mM AQMS at 2.0 M ionic strength
Long-Term Stability and Regeneration of V.cholerae DNA Biosensor
Figure 8 shows the shelf life of the V. cholerae DNA biosensor. The DNA biosensor showed the highest response to the detection of 5 μM of V. cholerae cDNA for the first month of the experimental period. The electrochemical DNA biosensor was able to retain 95% of its initial DPV current after 58 days of storage period. The DNA hybridisation response was then gradually decreased to about 75% of its original response on the 75th day and exhibited 40% of its initial performance on the 100th operational day. The bioactivity of the immobilised capture probe was finally declined to 30% after 3 months of storage period. The reproducibility of each calibration point, which was repeated on five replicate DNA electrodes, gave satisfactory relative standard deviation (RSD) between 2.4 and 4.5% (n =5).
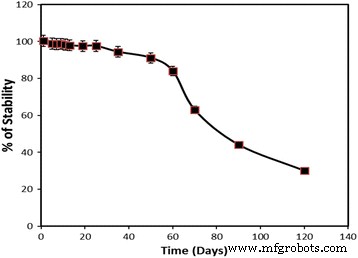
The life span profile of the fabricated V. cholerae DNA sensing electrode. The electrode was stored in 0.05 M potassium phosphate buffer (pH 7.0) at 4 °C after every DPV measurement was taken
Regeneration of biosensor indicates whether the biosensor is reusable for a series of consecutive analyses. The regeneration method used in this study was conducted based on previously reported protocol in other studies [11, 21] with slight modifications. In this study, 0.1 M of NaOH solution was used as the regeneration solution to break the hydrogen bonds between base pairs of hybridised dsDNA. With the result from Fig. 9, it is notable that the DNA biosensor response declined significantly after incubation in 0.1 M of NaOH and the percentage of the DNA biosensor response reduced from 35.1 to 5.2% relative to the DNA biosensor initial response after incubation in 0.1 M of NaOH solution from 30 to 240 s. The DNA biosensor response decreased with the increasing incubation time signifies the hydrogen bonds between hybridised dsDNA were broken up by the alkaline regeneration solution. However, rehybridisation of the DNA biosensor was able to attain almost 100% of its initial response for a consecutive six DNA analyses with a reversibility RSD of 5%.
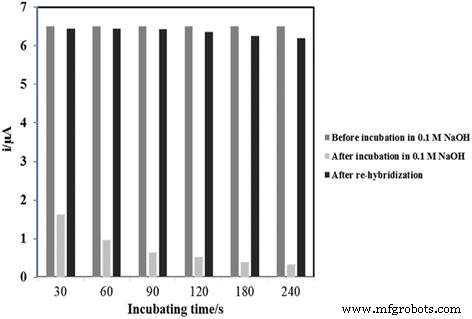
Repeatability of V. cholerae DNA biosensor using 0.1 M NaOH regeneration solution and rehybridization solution containing 5 μM cDNA and detection probe and 1 mM AQMS at 2.0 M ionic strength in 0.05 M potassium phosphate buffer (pH 7.0)
Determination of V. cholerae Bacteria with the Developed DNA Biosensor
The optimised DNA biosensor has been applied to quantify the V. cholerae DNA extracted from various V. cholerae bacterial strains. Table 3 presents the results acquired from DNA tests carried out with the hybridisation medium spiked with different strains of V. cholerae DNAs and other bacterial species at a concentration within the calibration range of the DNA biosensor. The DNA biosensor showed superior selectivity towards V. cholerae J3324–I, V. cholerae J3324–II, and V. cholerae UVC1324 with high DPV current response and low current signals were obtained for the evaluation of both Citrobacter freundii (CF-I) and Citrobacter freundii (CF-II).
Recovery of V. cholerae J3324 and V. cholerae UVC1324 DNAs at three different concentrations spiked into the hybridisation buffer demonstrated 91.4 ± 2.2% to 108.9 ± 4.8% (n =3) of recoveries percentage (Table 4). This result suggests that the proposed PSA-AuNPs-based electrochemical DNA biosensor could be adopted for highly reliable and accurate detection of V. cholerae DNA in environmental and clinical samples.
Performance Comparison with Other Reported V. cholerae DNA Biosensors
Based on the data summarised in Table 5, the proposed electrochemical DNA biosensor based on PSA-AuNPs immobilisation material shows an exceptional broad linear quantification range compared to other planar two-dimensional electrodes as the DNA supporters. This clearly demonstrates the advantage of the micro-sized latex particles where the polymeric PSA is capable to intensify the probe binding capacity with a simple loading method via the classical EDC/NHS coupling compared to avidin-biotin technology [24, 26] and ultra-low detection limit in zeptomolar range with reasonable assay time.
Conclusions
This study reports the development of an electrochemical DNA biosensor for the detection of one of the most devastating high-risk V. cholerae pathogens. The PSA-AuNPs-modified DNA biosensor can be used for direct detection of DNA of interest from the extracted DNA without the need of amplification reaction via conventional PCR method, which is commonly used in those previously reported V. cholerae DNA biosensors. In addition, no further dilution of the extracted DNA is needed as the high-capacity AuNPs-doped latex microspheres-based DNA biosensor is highly sensitive for the quantitation of DNA at extremely low level in sub zeptomolar range. Therefore, the electrochemical DNA biosensor is greatly suitable as a surveillance and diagnostic tool to control the epidemic of the fatal intestinal infection.
纳米材料
- 将视觉数据与物联网集成的潜力
- 用于灵敏快速检测卵巢癌细胞的柔性石墨烯生物传感器的演示
- 在 Au 纳米颗粒修饰的玻碳电极上增强光电化学检测尿酸
- 一种由亚克力-金纳米复合材料制成的高灵敏度电化学 DNA 生物传感器,用于确定龙鱼的性别
- 具有分层多孔结构的单分散碳纳米球作为超级电容器的电极材料
- 使用聚(4-苯乙烯磺酸-共-马来酸)增强金磁性纳米颗粒的稳定性:用于蛋白质检测的定制光学特性
- 控制合成不同形态的 BaYF5:Er3+、Yb3+ 以增强上转换发光
- 用于 CCRF-CEM 开启检测的基于氧化石墨烯的荧光适体传感器
- 具有高拉伸性的导电 TPU 纳米纤维复合材料用于柔性应变传感器
- 用于高速、室温直接太赫兹检测的大面积均匀石墨烯纳米网的制造
- 石胆酸修饰的金纳米粒子对肝癌细胞的凋亡作用
- Impossible Objects 与巴斯夫合作进行复合 3D 打印


